-
J Mol Evol. 2006 Jun;62(6):765-71. Epub 2006 Apr 28.
-
On the paucity of duplicated genes in Caenorhabditis elegans operons.
次に斜め読みするのはこの論文。
線虫の遺伝子重複がオペロン内の遺伝子とそうでない遺伝子で違うのか違わないのかという話。
えらい込み入った話だが、発想としてはおもしろい。
線虫の15%の遺伝子はオペロンにあるらしい。
その遺伝子が遺伝子重複がどれほど起きているか調べた。
すると、オペロン以外の遺伝子よりも重複している頻度が少なかったらしい。
オペロン遺伝子では下流の遺伝子がプロモーターを持ってないので、
それが分断されてしまうと困る。
それで、遺伝子重複が起こりにくくなっているというのが仮説。
遺伝子重複が起きた時期、重複の仕方、などを調べていくと仮説が正しいようなデーターがえられたそうだ。
内容としてはバイオインフォマティックスしかしてないな。
表しかない。
こういう論文は発想力で勝負なんだろうな。
解析結果も仮説どうりだったようだ。
こういう解析は結構勉強しないとできそうにもない。
それなりに評価されてもいいと思う。
しかし、こういう解析の仕方で危険なのは意外な結果がでた時には解釈不能。
解釈が不能という事はデーターとして扱えない。
つまり解釈できる事しか解析できないというのが危険だな。
疑問に思ったのが、なぜ遺伝子重複するのにプロモーターが壊れてはいけないのだろう。
この論文の重大な落ち度に気づいてしまった!
もともとオペロンだったが単独の遺伝子になってしまった場合が考慮されていない。
つまり、オペロン内の遺伝子は遺伝子重複が邪魔されているのではなくて、
遺伝子重複した遺伝子はオペロンから離脱しているということはないのだろうか。
PR
昨日の夜はサッカーがあってそのまま2時過ぎ位まで寝られなかった。
最近はそんな日がおおいなあ。
今日はこの論文を斜め読みからはじめるか。
Blumenthal T 昨日と一緒の著者。
続けて同じ著者のほうが読みやすいかな。
Blumenthal Tはトランススプライシングの分野で精力的にがんばっている人らしい。
今回の論文は、SLプロテインと新規に見つかった線虫のSL snRNPについての話。
SL75pとSL21pとそのパラログSL26pがある。
そのうちSL75pとSL21pがSL1のsnRNPコンプレックスに含まれている。
SL26pは含まれていない。
しかし、SL26pはSm Yという新規のsnRNAコンポーネントに含まれている。
SL75pをノックダウンすると致死なのだが、
SL26pとSL21pを個別にノックダウンしても致死にはならず、
SL26pとSL21pを両方ともノックダウンすると致死になる。
これは機能的にSL26pとSL21pがオーバーラップしているからという事を示唆している。
SL26pとSL75pは結合するので、
SL1 RNA complex と Sm Y complexが結合していると思われる。
結局のところ、SLトランススプライシングに線虫で見つかったSm Yコンプレックスが重要な働きをしているのではないかという事が言いたいらしい。
Sm ribonuclearproteins 7つの保存されたSmモチーフをもつタンパク質。SnRNPのコアになる。
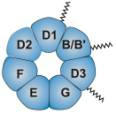
snRNPs
Birkhäuser Verlag, Basel, Switzerland) Oct 61(19-20), D. Schumperli and R. S. Pillai, The Special Sm Core Structure of the U7 snRNP: Far-reaching Significance of a Small Nulcear Ribonucleoprotein, pp2560-1570, 2004, PMID: 15526162.
U1
U2
U4
U5
最近はそんな日がおおいなあ。
今日はこの論文を斜め読みからはじめるか。
-
-
RNA. 2007 Apr;13(4):511-20. Epub 2007 Feb 5.
-
A novel family of C. elegans snRNPs contains proteins associated with trans-splicing.
Blumenthal T 昨日と一緒の著者。
続けて同じ著者のほうが読みやすいかな。
Blumenthal Tはトランススプライシングの分野で精力的にがんばっている人らしい。
今回の論文は、SLプロテインと新規に見つかった線虫のSL snRNPについての話。
SL75pとSL21pとそのパラログSL26pがある。
そのうちSL75pとSL21pがSL1のsnRNPコンプレックスに含まれている。
SL26pは含まれていない。
しかし、SL26pはSm Yという新規のsnRNAコンポーネントに含まれている。
SL75pをノックダウンすると致死なのだが、
SL26pとSL21pを個別にノックダウンしても致死にはならず、
SL26pとSL21pを両方ともノックダウンすると致死になる。
これは機能的にSL26pとSL21pがオーバーラップしているからという事を示唆している。
SL26pとSL75pは結合するので、
SL1 RNA complex と Sm Y complexが結合していると思われる。
結局のところ、SLトランススプライシングに線虫で見つかったSm Yコンプレックスが重要な働きをしているのではないかという事が言いたいらしい。
Sm ribonuclearproteins 7つの保存されたSmモチーフをもつタンパク質。SnRNPのコアになる。
snRNPs
Birkhäuser Verlag, Basel, Switzerland) Oct 61(19-20), D. Schumperli and R. S. Pillai, The Special Sm Core Structure of the U7 snRNP: Far-reaching Significance of a Small Nulcear Ribonucleoprotein, pp2560-1570, 2004, PMID: 15526162.
U1
U2
U4
U5
-
RNA. 2007 Sep;13(9):1409-26. Epub 2007 Jul 13.
-
C. elegans sequences that control trans-splicing and operon pre-mRNA processing.
この論文は線虫のオペロン遺伝子間の配列をバイオインフォマティックに解析して、重要と思われる配列を解析している。
バイオインフォマティックで解析している部分はまったく持って理解不能。
よって分るところだけメモしとく。
線虫にはSL1とSL2という二つのSLRNAがある。
(ほやには一つしかない。)
SL2は主にオペロンの下流の遺伝子のトランススプライシングを行う。
SL1はどこでもいい。
オペロンの遺伝子間のプロセシングが行われるには以下の因子が必要。
CPSF Cleavage Polyadenylation Specificity
CstF Cleavage stimulation factor
SL2 snRNP SL2 small nuclear ribonuclear protein
Ur element
U-rich element
PAS (AAUAAA) poly adenylation signal
+++++++++++++++++++++++++++++++++++++++++
Common SL2 (operon) processing model
普通のオペロン遺伝子間のプロセシング。
Common SL1 processing model
SL1だけのトランススプライシング。
今回の解析でOuエレメントという保存された配列が初めて見つかった。OuエレメントはUrエレメントに似ているので同じ起源の配列である事がしさされる。しかし、UrエレメントにはCstfが必要。
Large separation operon processing model
オペロン遺伝子間の距離が長い場合、SL1とSL2のトランススプライシングが競合すると考えられる。
この図のほかにも、内部プロモーターが働いて、別々に転写されている事も考えられる。
SL1-TYPE Operon processing model
オペロン遺伝子間のスペースがないタイプのプロセシング。
ホヤのオペロンの大部分ははこのタイプらしいのでなんて書かれているか興味深い。
ポリA付加かトランススプライシングの競合が起こる。
ポリAが先に付加されると下流遺伝子のトランススプライシングは起こりえない。
下流のトランススプライシングが先に起こると、上流の遺伝子は分解されるか、ポリA付加される。
ただし、上流の遺伝子がポリA付加されるには、SLトランススプライシングによってふかされたブランチが取り除かれなければならない。
